I processen med at udføre genetiske undersøgelser støder vi ofte på utilstrækkelige RNA-prøver, for eksempel til at studere bittesmå anatomiske orale tumorer, endda enkeltcelleprøver og prøver af specifikke genmutationer, der transskriberes i meget lave niveauer i humane celler.For COVID-19-testen vil prøvestørrelsen selvfølgelig være meget lav, hvis podningen ikke er på det rigtige sted eller ikke er nok gange under prøvetagningen, hvilket er grunden til, at Kommissionen for Sundhed og Familieplanlægning udkom for to dage siden og bestod testen, og hvis nukleinsyreprøvetageren ikke tog seks prøver, kan du rapportere det.
Følsomheden af reagenset er vigtig, fordi vi har dette eller det problem, så hvad kan vi gøre for at forbedre følsomheden af RT-PCR?
Før vi diskuterer mulige løsninger, lad os nævne to store komplikationer med den situation, vi lige har nævnt.
Først og fremmest bekymrer vi os om RNA-tab, når vi kun har nogle få cellepopulationer i vores prøve.Hvis traditionelle separations- og rensningsmetoder anvendes, såsom kolonnemetode eller nukleinsyrepræcipitationsmetode, er der stor mulighed for, at de få prøver går tabt.En løsning er at tilføje et bærermolekyle, såsom tRNA, men selv da er der ingen garanti for, at vores genopretningseksperiment er OK.
Så hvad er en bedre måde?En god mulighed for dyrkede celler eller mikroanatomiske prøver er at bruge direkte lysis.
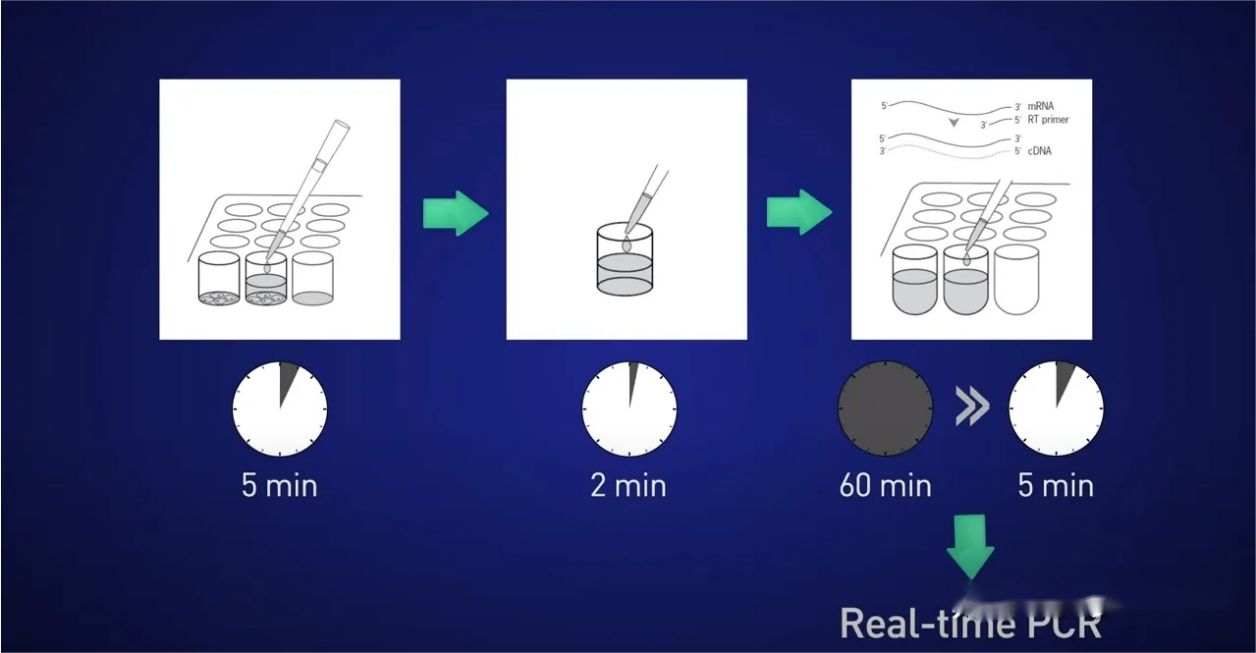
Ideen er at opdele cellerne i 5 minutter, frigive RNA i opløsningen, derefter stoppe reaktionen i 2 minutter, derefter tilføje lysatet direkte til den omvendte transkriptionsreaktion, så intet RNA går tabt, og til sidst placere det resulterende cDNA direkte ind i realtidsreaktionen.
Men hvad nu hvis vi på grund af et begrænset udgangspunkt eller en lille mængde målgenekspression kan genbruge alt RNA og stadig ikke levere nok skabeloner til at få et godt realtidssignal?
I dette tilfælde kan præ-amplifikationstrinnet være meget nyttigt.
Det følgende er et skema til at øge følsomheden efter omvendt transkription.Før vi starter, skal vi spørge nedstrøms, hvilke mål vi er interesserede i, for at designe specifikke primere til disse mål til præ-amplifikation.
Dette kan opnås ved at skabe en blandet primer med op til 100 par primere og en reaktionscyklus på 10 til 14 gange.Derfor er en Master Mix, der er specielt designet til dette krav, nødvendig for at præamplificere det opnåede cDNA.
Begrundelsen for at sætte antallet af cyklusser mellem 10 og 14 er, at dette begrænsede antal cyklusser sikrer tilfældighed mellem de forskellige mål, hvilket er afgørende for forskere, der har behov for kvantitativ molekylær information.
Efter præ-amplifikation kan vi få en stor mængde cDNA, så detektionsfølsomheden i back-end er meget forbedret, og vi kan endda fortynde prøven og udføre flere real-time PCR-reaktioner for at eliminere mulige tilfældige fejl.
Indlægstid: 11-apr-2023